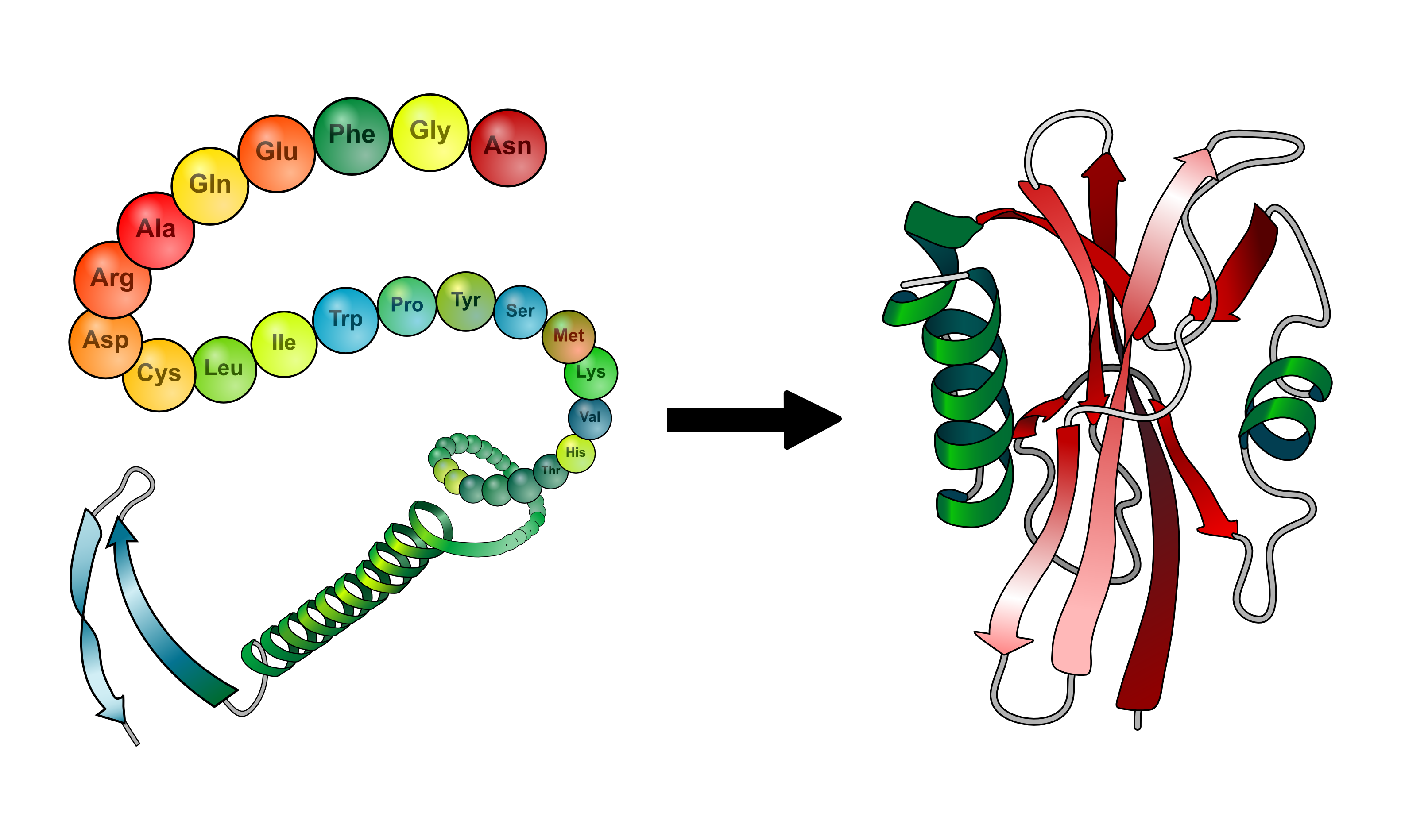
Blood oxygenation level-dependent (BOLD) signal
BOLD signal BOLD fMRI is often used to measure the hemodynamic response resulting from changes in the oxygenation level in the blood vessels. Yet to be explained is the relationship between neuronal activity and the measured fMRI signal. In a seminal paper, Ogawa et al., (1990) showed that blood-oxygenation-level-dependent (BOLD) constitutesRead More →